Un juego de ordenador para científicos
El Instituto Howard Huges desarrolla un programa 'online' para diseñar nuevas proteínas
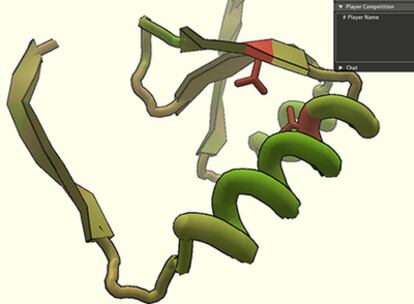
¿Quién ha dicho que los juegos de ordenador son una pérdida de tiempo? No se trata ya de que con el Tetris se mejore la visión espacial o que una buena partida al World or Warcraft ayude al pensamiento lógico. Los investigadores del Instituto Médico Howard Hughes de la Universidad de Washington han creado un juego online que consiste en plegar moléculas para formar diversas proteínas.
El juego recibe el nombre de Foldit! (¡Pliegalo!) es gratuito y a través de la red pueden participar personas de todo el mundo desde ciudadanos anónimos a grupos de grandes universidades como Berkeley. Para descargárselo hay que visitar Foldit!. Su objetivo es diseñar nuevas proteínas que podrían servir para crear nuevos medicamentos, aunque de momento los participantes prueban a componer las que ya se conocen.
La idea surgió como una prolongación de la investigación de David Baker, para comprender la forma en la que las proteínas (los ladrillos de construcción de las células) se pliegan en formas tridimensionales únicas. La predicción de las formas que tomarán las proteínas naturales es uno de los desafíos preeminentes de la biología, y el modelado de incluso una proteína pequeña requiere de la fabricación de trillones de cálculos. Durante los últimos tres años, voluntarios de todo el mundo (casi 200.000 personas) se han prestado a formar una red de ordenadores (Rosetta@home) para poder realizar esos cálculos.
Sin embargo, ni siquiera así conseguían dar con los calculos. "¡La gente escribía diciendo: `¡Oigan! ¡La computadora está haciendo cosas tontas! Sería fantástico si pudiéramos ayudar a dirigirla'", recuerda Baker en la web del Instituto, quien se encuentra en la Universidad de Washington (UW) donde desarrolló la red Rosetta y el algoritmo que usa para tratar de hallar las soluciones.
El hombre en ayuda de la máquina
Al contrario de lo que suele suceder, se pensó que el hombre podría ahora ayudar a los ordenadores a conseguir dar con una solución. Baker cuenta en la web del Instituto Hughes que no sabía cómo podría lograrlo hasta hace aproximadamente 18 meses, cuando fue de excursión al Monte Rainier con su vecino David Salesin, científico informático de la Universidad de Washington. Juntos buscaban una forma de hacer que Rosetta fuera más interactiva. Con un gusto inherente por la competición, Salesin pensó que un juego en Internet de múltiples jugadores era la forma de lograrlo. Para cuando volvieron de nuevo al coche, ya se habían decidido por esa idea. Salesin le dio a Baker los nombres de tres colegas, liderados por el científico computacional de UW Zoran Popovi?, que podrían ayudar a Baker a crear el juego.
Durante varios meses después, Popovi? y sus estudiantes Adrien Treuille y Seth Cooper, crearon el programa, y el equipo lo probó en pequeña escala. Un juego entre los equipos de la Universidad de California y de la Universidad de Illinois despertó fervor y vítores inesperados entre los espectadores. "Participaron 30 ó 40 personas", dice Baker. "La competición fue muy intensa".
Un juego para todos
Foldit! lleva a los jugadores a través de una serie de niveles de práctica diseñados para enseñar los fundamentos de cómo se pliegan las proteínas, antes de darles rienda suelta con proteínas verdaderas de la naturaleza. En la web del Instituto cuenta como "Nuestra meta principal era asegurarnos de que cualquier persona podría hacerlo, aunque no supieran qué era la bioquímica o el plegamiento proteico", dice Popovi?. Por el momento, el juego sólo utiliza sustancias cuyas estructuras tridimensionales han sido resueltas por los investigadores. Pero, Popovi? dice, "pronto introduciremos rompecabezas para los cuales no sabemos la solución".
Baker tiene mucha esperanza de que el juego acelere el a veces aburrido trabajo de la predicción de estructuras. Pero la parte del juego que lo entusiasma más hará su debut el próximo otoño, cuando los jugadores podrán diseñar proteínas completamente nuevas. Baker piensa que, con la habilidad de cualquier persona con una computadora y una conexión a Internet, el ritmo de descubrimiento podría aumentar vertiginosamente. "Mi sueño es que un niño de 12 años de Indonesia resulte ser un prodigio y construya una curación para el VIH", afirma.
Tu suscripción se está usando en otro dispositivo
¿Quieres añadir otro usuario a tu suscripción?
Si continúas leyendo en este dispositivo, no se podrá leer en el otro.
FlechaTu suscripción se está usando en otro dispositivo y solo puedes acceder a EL PAÍS desde un dispositivo a la vez.
Si quieres compartir tu cuenta, cambia tu suscripción a la modalidad Premium, así podrás añadir otro usuario. Cada uno accederá con su propia cuenta de email, lo que os permitirá personalizar vuestra experiencia en EL PAÍS.
¿Tienes una suscripción de empresa? Accede aquí para contratar más cuentas.
En el caso de no saber quién está usando tu cuenta, te recomendamos cambiar tu contraseña aquí.
Si decides continuar compartiendo tu cuenta, este mensaje se mostrará en tu dispositivo y en el de la otra persona que está usando tu cuenta de forma indefinida, afectando a tu experiencia de lectura. Puedes consultar aquí los términos y condiciones de la suscripción digital.




























































